HITOC
HITOC répond à une limite majeure des modèles in vitro actuels de l’intestin humain: l’absence de systèmes physiologiquement segmentés et intégrant un système nerveux entérique (SNE) fonctionnel, capables de reproduire la complexité structurale, fonctionnelle et électrophysiologique du tractus digestif. Bien que les technologies d’organoïdes et d’organes-sur-puce (OoC) aient transformé la modélisation des tissus humains, les dispositifs existants restent en grande partie confinés à un seul segment intestinal et ne prennent pas en compte des compartiments clés tels que le SNE. Ce manque limite considérablement l’étude des interactions hôte-pathogène selon le segment, des signaux microbiens, et de l’axe intestin-cerveau, qui dépendent intrinsèquement de la régionalisation et de l’activité neuronale locale.
Coordinateur

Chargé de recherche
Inserm
Institutions et établissements impliqués
Work Packages
CNRS; Inserm; Aix Marseille Université; Institut Pasteur de Lille; Institut Pasteur de Paris
Les objectifs scientifiques sont structurés autour de Work Packages :
- WP1 : standardisation des protocoles hiPSC et de l’analyse des données dans une démarche de science ouverte ;
- WP2 : développement d’un intestin sur puce inspiré du développement, mimant des segments intestinaux spécifiques ;
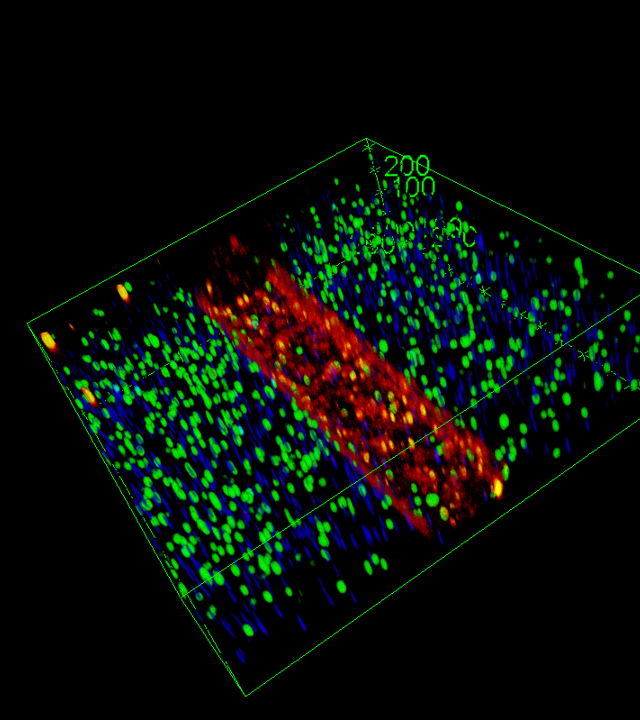
- WP3 : intégration fonctionnelle et suivi de l’épithélium intestinal et des neurones à l’aide de réseaux d’électrodes multicanaux (MEA) 3D transparents ;
- WP4 : validation et applications translationnelles des modèles HITOC.
Programme de recherche
Pour répondre à ces verrous, HITOC propose une nouvelle génération de système intestinal sur puce dérivé de cellules souches pluripotentes induites humaines (hiPSC), conçu pour reproduire la segmentation rostro-caudale du tube digestif (duodénum, jéjunum, iléon, côlon) et son intégration fonctionnelle avec des neurones entériques. Notre approche s’inspire de la biologie du développement, en utilisant des gradients morphogénétiques spatialisés et une micro-ingénierie de confinement pour guider la différenciation segmentaire directement sur puce. Une innovation majeure du projet réside dans l’intégration d’un compartiment SNE fonctionnel, dérivé de cellules de crête neurale vagale ou de progéniteurs entériques, pendant la phase de maturation tissulaire.
Cette interface neuro-épithéliale sera monitorée en temps réel grâce à des microélectrodes transparentes tridimensionnelles (MEA) intégrées au dispositif, permettant l’enregistrement électrophysiologique de l’activité neuronale, l’imagerie calcique et le suivi de l’intégrité de la barrière épithéliale par impédancemétrie (ECIS). Ces capteurs multi-paramétriques, non invasifs et en temps réel, offrent une lecture fonctionnelle sans précédent de l’intestin humain. À titre de validation, le système HITOC sera utilisé pour étudier comment des pathogènes humains restreints, tels que Shigella et les Norovirus—connus pour leur tropisme intestinal spécifique— altèrent l’intégrité de l’épithélium et le fonctionnement du SNE. En parallèle, nous analyserons les effets de métabolites microbiens, notamment les acides gras à chaîne courte (AGCC), sur les circuits sensoriels intestinaux et l’activité neuronale entérique, dans le but de mieux comprendre les mécanismes de l’axe intestin-cerveau.

Résultats attendus
En combinant auto-organisation des organoïdes, microfabrication et analyse fonctionnelle, HITOC établit un nouveau standard pour la modélisation intestinale humaine et offrira un outil de référence pour l’étude des infections, des interactions hôte-microbiote et des troubles neurogastro-intestinaux dans un contexte préclinique entièrement humain.
Le consortium
HITOC réunit un consortium français pluridisciplinaire, aux expertises complémentaires en biologie des hiPSC, développement du SNE, bio-ingénierie microfluidique, électrophysiologie et biologie des infections. Il s’appuie sur des partenaires de l’Inserm, du CNRS et des Instituts Pasteur de Paris et de Lille, avec une solide expérience collaborative et technologique. Le modèle sera modulaire, interopérable et standardisé de manière ouverte pour assurer une diffusion large et s’aligner pleinement avec les objectifs du PEPR MED-OOC.
CIIL ; IEMN ; IBDM ; TENS ; Plate-Forme Technologique Biomatériaux et Microfluidiques
Plus de projets